NEWS
NEWS
Single cell sequencing opens new avenues for eradicating leukaemia at its source
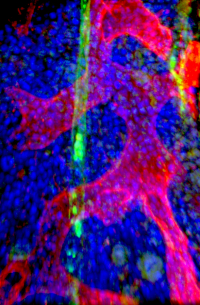
Stem cells (blue) in bone marrow together with a sinusoidal blood vessel (red) and an arteriolar blood vessel. Credit: Jude Al-Sabah/DKFZ
A new method, described in a study published today in the journal Nature Communications, has the potential to boost international research efforts to find drugs that eradicate cancer at its source.
Most cancerous tissue consists of rapidly dividing cells with a limited capacity for self-renewal, meaning that the bulk of cells stop reproducing after a certain number of divisions. However, cancer stem cells can replicate indefinitely, fuelling long-term cancer growth and driving relapse.
Cancer stem cells that elude conventional treatments like chemotherapy are one of the reasons patients initially enter remission but relapse soon after. In acute myeloid leukaemia, a form of blood cancer, the high probability of relapse means fewer than 15% of elderly patients live longer than five years.
However, cancer stem cells are difficult to isolate and study because of their low abundance and similarity to other stem cells, hampering international research efforts in developing precision treatments that target malignant cells while sparing healthy ones.
Researchers from the Centre for Genomic Regulation (CRG) and the European Molecular Biology Laboratory (EMBL) have overcome this problem by creating MutaSeq, a method that can be used to distinguish cancer stem cells, mature cancer cells and otherwise healthy stem cells based on their genetics and gene expression.
“RNA provides vital information for human health. For example, PCR tests for coronavirus detect its RNA to diagnose COVID-19. Subsequent sequencing can determine the virus variant,” explains Lars Velten, Group Leader at the CRG and author of the paper. “MutaSeq works like a PCR test for coronavirus, but at a much more complex level and with a single cell as starting material.”
To determine if a single cell is a stem cell, the researchers used MutaSeq to measure thousands of RNAs at the same time. To then find out if the cell is cancerous or healthy, the researchers carried out additional sequencing and looked for mutations. The resulting data helped researchers track if stems cells are cancerous or healthy and helped determine what makes the cancer stem cells different.
“There are a huge number of small molecule drugs out there with demonstrated clinical safety, but deciding which cancers and more specifically which patients these drugs are well suited for is a daunting task,” says Lars Steinmetz, Professor at Stanford University, Group Leader at EMBL Heidelberg and author of the paper. “Our method can identify drug targets that might not have been tested in the right context. These tests will need to be carried out in controlled clinical studies, but knowing what to try is an important first step.”
The method is based on single cell sequencing, an increasingly common technique that helps researchers gather and interpret genome-wide information from thousands of individual cells. Single cell sequencing provides a highly detailed molecular profile of complex tissues and cancers, opening new avenues for research.
Explaining their next steps, Lars Velten says:
“We have now brought together clinical researchers from Germany and Spain to apply this method in much larger clinical studies. We are also making the method much more streamlined. Our vision is to identify cancer stem cell specific drug targets in a personalized manner, making it ultimately as easy for patients and doctors to look for these treatments as it is testing for coronavirus”.
La secuenciación de células individuales abre nuevas vías para erradicar la leucemia en su origen
Un nuevo método, descrito en un estudio publicado hoy en la revista Nature Communications, tiene el potencial para impulsar nuevos esfuerzos de investigación internacionales para encontrar fármacos que eliminen el cáncer en su origen.
La mayor parte de un tejido canceroso consiste en células que tienen una capacidad limitada de renovación, lo que implica que dichas células dejan de reproducirse tras un determinado número de divisiones. Sin embargo, las células madre cancerosas pueden replicarse indefinidamente, lo que impulsa el crecimiento del cáncer a largo plazo y provoca la recaída.
Las células madre cancerosas, que eluden los tratamientos convencionales como la quimioterapia, son una de las razones por las que los pacientes entran en remisión al principio, pero sufren una recaída después. En la leucemia mieloide aguda (un tipo de cáncer de la sangre conocido como AML por sus siglas en inglés), la alta probabilidad de recaída significa que poco menos del 15% de los pacientes de edad avanzada tienen una esperanza de vida de más de cinco años.
Las células madre cancerosas son difíciles de aislar y estudiar debido a su baja abundancia y similitud con otras células madre. La rareza de estas células es un gran escollo para que los equipos de investigación a nivel mundial puedan desarrollar nuevos tratamientos de precisión que eliminen las células malignas sin afectar a las sanas.
Un equipo científico del Centro de Regulación Genómica (CRG), en colaboración con el Laboratorio Europeo de Biología Molecular (EMBL) en Heidelberg (Alemania), ha dejado atrás este problema con la creación de MutaSeq, un método que puede usarse para distinguir entre las células madre cancerosas, las células cancerosas maduras y las células madre sanas, en función de su genética y expresión génica.
“El ARN proporciona información vital para la salud humana. Por ejemplo, las pruebas de PCR para el coronavirus detectan el ARN para diagnosticar la COVID-19. La secuenciación posterior puede determinar la variante del virus,” explica Lars Velten, jefe de grupo del CRG y autor del artículo. “MutaSeq funciona como una prueba PCR para el coronavirus, pero a un nivel mucho más complejo y usando una célula individual como punto de partida.”
Para determinar si una célula individual es una célula madre, el equipo científico utilizó MutaSeq para medir miles de moléculas de ARN al mismo tiempo. El equipo llevó a cabo una secuenciación adicional en busca de mutaciones que pudieran diferenciar las células madres cancerosas de las sanas. Los resultados ayudaron a monitorizar si las células madre eran cancerosas o sanas, y a determinar qué hace que las células madre cancerosas sean diferentes.
“Existe una gran cantidad de pequeñas moléculas farmacológicas con una seguridad clínica demostrada, pero es una tarea abrumadora decidir para qué cánceres y más específicamente para qué pacientes son”, afirma Lars Steinmetz, profesor de la Universidad de Stanford, jefe de grupo en el EMBL Heidelberg y co-autor del estudio. “Nuestro método puede identificar nuevas dianas terapéuticas para fármacos que no han sido probados en el contexto adecuado. Estas pruebas deberán realizarse en el marco de estudios clínicos controlados, pero saber qué probar es el primer paso.”
El método se basa en la secuenciación del ARN de una célula individual (conocida internacionalmente como scRNA-seq), una técnica cada vez más común que ayuda a los investigadores a recopilar e interpretar información de todo el genoma de miles de células individuales a la vez. La secuenciación de células individuales proporciona un perfil molecular muy detallado de tejidos complejos y cánceres, que a su vez abre nuevas vías para la investigación.
Lars Velten explica los próximos pasos de su investigación, concluyendo:
“Hemos reunido a equipos de investigación clínica de Alemania y España para aplicar este método en estudios clínicos mucho más amplios. También estamos simplificando mucho el método. Nuestra visión a largo plazo es identificar nuevas dianas terapéuticas en las células madre cancerosas de forma personalizada, haciendo que para los pacientes y los médicos sea tan fácil buscar estos tratamientos como realizar una prueba de diagnóstico para el coronavirus”.
EN CATALÀ
La seqüenciació de cèl·lules individuals obre noves vies per a erradicar la leucèmia en el seu origen
Un nou mètode, descrit en un estudi publicat avui a la revista Nature Communications, té el potencial per impulsar nous esforços de recerca internacionals per a trobar fàrmacs que eliminin el càncer en el seu origen.
La major part d’un teixit cancerós consisteix en cèl·lules que tenen una capacitat limitada de renovació, fet que implica que les esmentades cèl·lules deixen de reproduir-se després d’un nombre determinat de divisions. Tanmateix, les cèl·lules mare canceroses poden replicar-se indefinidament i això impulsa el creixement del càncer a llarg termini i provoca la recaiguda.
Les cèl·lules mare canceroses, que eludeixen els tractaments convencionals com la quimioteràpia, són una de les raons per les quals els pacients entren en remissió a l’inici, però pateixen una recaiguda després. En la leucèmia mieloide aguda (un tipus de càncer de la sang conegut com a AML per les seves sigles en anglès), l’alta probabilitat de recaiguda significa que poc menys del 15% dels pacients d’edat avançada tenen una esperança de vida de més de cinc anys.
Les cèl·lules mare canceroses són difícils d’aïllar i estudiar perquè són poc abundants i per la seva similitud amb d’altres cèl·lules mare. La raresa d’aquestes cèl·lules és un gran escull per a què els equips de recerca a nivell mundial puguin desenvolupar nous tractaments de precisió que eliminin les cèl·lules malignes sense afectar les sanes.
Un equip científic del Centre de Regulació Genòmica (CRG), en col·laboració amb el Laboratori Europeu de Biologia Molecular (EMBL) a Heidelberg (Alemanya), ha deixat enrere aquest problema amb la creació de MutaSeq, un mètode que pot emprar-se per distingir entre les cèl·lules mare canceroses, les cèl·lules canceroses madures i les cèl·lules mare sanes, en funció de la seva genètica i expressió gènica.
“L’ARN proporciona informació vital per a la salut humana. Per exemple, les proves de PCR pel coronavirus detecten l’ARN per a diagnosticar la COVID-19. La seqüenciació posterior pot determinar la variant del virus,” explica Lars Velten, cap de grup del CRG i autor de l’article. “MutaSeq funciona com una prova PCR pel coronavirus, però a un nivell molt més complex i emprant una cèl·lula individual com a punt de partida.”
Per determinar si una cèl·lula individual és una cèl·lula mare, l’equip científic utilitzà MutaSeq per a mesurar milers de molècules d’ARN a la vegada. L’equip va dur a terme una seqüenciació addicional per cercar mutacions que poguessin diferenciar les cèl·lules mare canceroses de les sanes. Els resultats ajudaren a monitoritzar si les cèl·lules mare eren canceroses o sanes, i a determinar què fa que les cèl·lules mare canceroses siguin diferents.
“Existeix una gran quantitat de petites molècules farmacològiques amb una seguretat clínica demostrada, però és una tasca aclaparadora decidir per a quins càncers i més específicament per a quins pacients són”, afirma Lars Steinmetz, professor de la Universitat de Stanford, cap de grup a l’EMBL Heidelberg i co-autor de l’estudi. “El nostre mètode pot identificar noves dianes terapèutiques per a fàrmacs que no han estat provats en el context adequat. Aquestes proves hauran de realitzar-se en el marc d’estudis clínics controlats, però saber què provar és el primer pas.”
El mètode es basa en la seqüenciació de l’ARN d’una cèl·lula individual (coneguda internacionalment com a scRNA-seq), una tècnica cada cop més comuna que ajuda els investigadors a recopilar i interpretar informació de tot el genoma de milers de cèl·lules individuals a la vegada. La seqüenciació de cèl·lules individuals proporciona un perfil molecular molt detallat de teixits complexos i càncers, que alhora obre noves vies per a la recerca.
Lars Velten explica els propers passos de la seva recerca, concloent:
“Hem reunit a equips de recerca clínica d’Alemanya i Espanya per aplicar aquest mètode en estudis clínics molt més amplis. També estem simplificant molt el mètode. La nostra visió a llarg termini és identificar noves dianes terapèutiques en les cèl·lules mare canceroses de manera personalitzada, fent que per als pacients i els metges sigui tan fàcil buscar aquests tractaments com realitzar una prova de diagnòstic pel coronavirus.”